
COMPRENDRE LA RÉPONSE IMMUNITAIRE AU SRAS-COV-2
Charu Kaushic | 6 juillet 2020
Télécharger l'article au complet
Charu Kaushic, M. Sc. Ph.D. Directrice scientifique, Institut des maladies infectieuses et Immunitaires (IMII) des Instituts de recherche en santé du Canada (IRSC), professeure, Département de pathologie et de médecine moléculaire, Centre de recherche en immunologie de l’Université McMaster, Hamilton, Canada.
Résumé
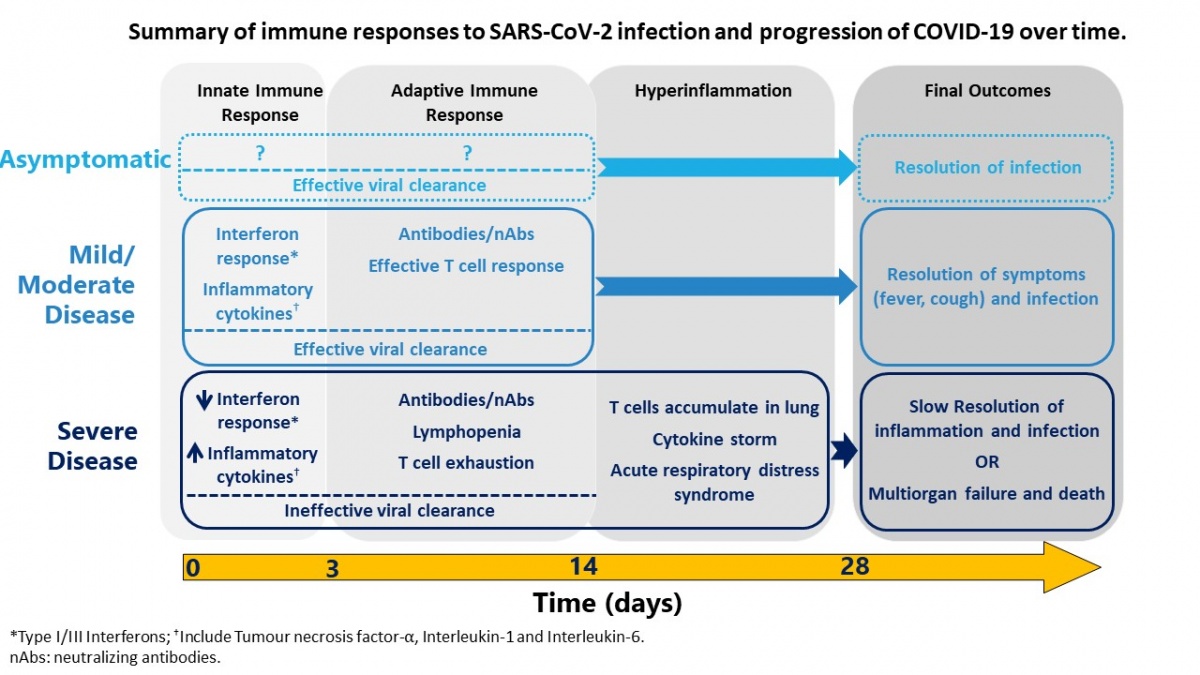
Le coronavirus du syndrome respiratoire aigu sévère 2 (SRAS-CoV-2) est un coronavirus d’émergence récente hautement infectieux qui cause la maladie à coronavirus 2019 (COVID-19). Cette dernière se caractérise principalement par des symptômes respiratoires légers à graves pouvant nécessiter une hospitalisation et, dans environ 1 % des cas, provoquer la mort. Des tests diagnostiques pour la détection du virus dans les voies respiratoires ont rapidement été mis au point, quelques semaines après l’éclosion, et ont joué un rôle important dans les stratégies de santé publique à l’échelle mondiale pour dépister, suivre et isoler les cas, ce qui a probablement sauvé des millions de vies, partout dans le monde. Cependant, il faudra faire encore beaucoup de recherche pour comprendre ce nouveau virus, la maladie, ainsi que la réponse immunitaire qui pourrait protéger de la COVID-19 ou y contribuer. À la lumière des données qui émergent rapidement, l’infection par le SRAS-CoV-2 présente une symptomatologie différentielle, qui serait probablement le résultat de l’interaction entre le virus et la réponse immunitaire de l’hôte (Figure). Les issues les plus graves de la maladie sont corrélées à une réponse immunitaire excessive. On ne comprend pas encore entièrement le rôle des anticorps dans la protection immunitaire, ce qui rend les prédictions concernant l’immunité collective incertaines et l’utilité des « certificats d’immunité » difficile à évaluer. L’ultime stratégie de sortie de la pandémie actuelle comportera très probablement une combinaison de thérapies efficaces pour réduire le fardeau de la maladie et de vaccins pour prévenir la transmission.
1. Réponses immunitaires aux virus : immunité innée et immunité acquise
Les virus sont des parasites inframicroscopiques qui ont besoin de cellules hôtes pour se répliquer. Ces minuscules organismes sont composés d’informations génétiques fondamentales sous forme d’acides nucléiques (ARN ou ADN) encapsulés dans une membrane protéique appelée « capside ». De nombreux virus comportent également une capsule externe, l’enveloppe, qui est une membrane lipidique contenant des glycoprotéines virales qui facilitent l’entrée du virus dans les cellules hôtes. La principale fonction de l’ensemble de cette structure virale est d’infecter les cellules, de prendre le contrôle de la machinerie de la cellule hôte pour se multiplier et produire de grandes quantités de virions, lesquels sont ensuite libérés et répètent le processus dans d’autres cellules. Alors que les virus peuvent infecter une grande variété d’animaux et de plantes, la plupart s’attaquent seulement à une variété étroite d’hôtes, et peu d’entre eux peuvent infecter différentes espèces. Les coronavirus font partie de la dernière catégorie1. Lors de l’infection, l’hôte détecte le virus, l’identifie comme une entité « étrangère » et organise une défense par l’intermédiaire de son système immunitaire2. Le système immunitaire humain est un réseau complexe de cellules et d’organes qui peuvent détecter les organismes nuisibles et se défendre contre eux au moyen de divers mécanismes.
La réponse immunitaire de l’humain à un virus se divise en différentes phases, qui s’amorcent dans les minutes suivant l’infection virale et durent des semaines, des mois ou, parfois, des années3. La phase aiguë de la réponse immunitaire dure environ 72 heures; elle est dominée par « l’immunité innée », qui repose sur des cellules et des processus pouvant être déployés et agir rapidement afin de contenir le virus et de stopper sa réplication au sein de l’hôte. Chaque cellule du corps humain est équipée pour reconnaître les virus et y réagir, particulièrement les cellules qui tapissent les muqueuses, soit les parois humides de l’intérieur du corps qui recouvrent les voies respiratoires, le tractus gastro-intestinal, les voies urogénitales et d’autres organes. Lorsqu’un virus respiratoire, tel qu’un coronavirus, rencontre les cellules épithéliales qui tapissent les voies respiratoires, il se lie à des récepteurs spécifiques à la surface de la cellule hôte au moyen de son enveloppe de glycoprotéines virales. Cette liaison facilite l’entrée du virus dans la cellule, où il peut commencer à se répliquer, en prenant le contrôle de la machinerie cellulaire. Au même moment, divers récepteurs présents à la surface et dans le cytoplasme de la cellule hôte, appelés « récepteurs de reconnaissance de motifs moléculaires » (PRR, pour pattern recognition receptors), reconnaissent le virus et déclenchent la réponse immunitaire innée. Les mécanismes de cette réponse comprennent la production d’interféron (IFN) de types I et III et de facteurs cellulaires qui ont des effets antiviraux, ainsi que de facteurs inflammatoires qui transmettent des signaux d’alarme et envoient des cellules immunitaires, comme les macrophages, les cellules dendritiques et les cellules tueuses naturelles (NK, pour natural killer), au foyer d’infection. Le milieu antiviral et inflammatoire ainsi que les cellules immunitaires qui constituent la réponse immunitaire innée visent principalement a) à détruire le virus ou à restreindre sa réplication et b) à activer la phase suivante de la réponse immunitaire, dans les 48 à 72 heures qui suivent. La deuxième phase de la réponse immunitaire concerne l’immunité acquise (ou « adaptative »), qui dure pendant des semaines, voire des années, et à laquelle participent des organes et des tissus particuliers, comme les nœuds (ganglions) lymphatiques et des cellules immunitaires spécialisées, tels les lymphocytes B et T. La réponse immunitaire acquise est plus lente, mais peut ajuster avec une spécificité élevée les réactions immunitaires à une grande variété de pathogènes, comme des virus et des bactéries nuisibles. La réponse immunitaire acquise est capable de cibler de nouveaux virus que l’hôte n’a jamais rencontrés; elle est également responsable de la « mémoire » du système immunitaire qui permet à l’hôte de répondre beaucoup plus rapidement à des réinfections, même si elles se produisent des années plus tard.
Les vaccins nous permettent de tirer avantage de cette capacité à personnaliser une réponse secondaire spécifique, au moyen de virus affaiblis ou de fragments de virus; ainsi, lorsque l’hôte rencontre un virus pleinement infectieux, l’organisme peut lancer une réponse immunitaire secondaire rapide qui l’aide à résister à l’infection. La réponse immunitaire acquise comporte deux principaux éléments, soit 1) une immunité cellulaire constituée de lymphocytes T qui peuvent directement ou indirectement attaquer et tuer les cellules infectées et 2) une immunité humorale médiée par les lymphocytes B et les anticorps, des protéines complexes sécrétées par les cellules plasmatiques (lymphocytes B différenciés). Les anticorps sont présents dans le sang et les liquides organiques; ils exercent diverses fonctions, comme l’inhibition de la liaison virale, la neutralisation du virus ou l’activation des cellules immunitaires pour tuer le virus ou les cellules infectées. La caractéristique des anticorps réside dans leur capacité à reconnaître des épitopes très spécifiques du virus, ou « antigènes », et à s’y lier. Bien que les anticorps ne survivent que quelques jours à quelques semaines dans la circulation sanguine, les cellules plasmatiques peuvent continuer à en produire pendant des mois. Il existe cinq classes d’anticorps, soit les IgD, les IgM, les IgA, les IgE et les IgG, selon le type de chaîne protéique qui les constitue. Ils ont tous des structures, des caractéristiques et des fonctions légèrement différentes. Les anticorps IgM, IgG et IgA ont tous des fonctions antivirales, et les IgG sont ceux qui durent le plus longtemps; ils sont présents en grande quantité dans le sang et les liquides organiques et exercent une grande variété d’activités antivirales.
En résumé, le système immunitaire humain répond aux virus de différentes façons, en commençant par l’immunité innée, qui lance des réponses rapides et larges, suivie de l’immunité acquise, qui est médiée par des cellules immunitaires spécialisées et des anticorps et caractérisée par sa spécificité, sa diversité et sa mémoire. L’issue d’une exposition à un virus est déterminée par le rapport entre la capacité du virus à perturber et à vaincre la réponse de l’hôte et la capacité de l’hôte à lancer une réponse immunitaire antivirale rapide et puissante.
2. La réponse immunitaire au SRAS-CoV-2 : état actuel des connaissances
Les coronavirus appartiennent à une vaste famille de virus à ARN qui peuvent infecter plusieurs espèces de mammifères, y compris le chat, la chauve-souris, le chameau, la vache, le porc et l’humain4. Il existe sept espèces de coronavirus capables d’infecter les humains; ils causent tous des infections respiratoires. Quatre d’entre eux causent des rhumes (les alphacoronavirus 229E et NL3 et les bêtacoronavirus OC43 et HK41) dont la gravité varie, mais qui sont rarement mortels. Trois coronavirus qui causent des infections respiratoires beaucoup plus graves et ont des taux de mortalité élevée5,6 sont passés d’autres espèces aux humains au cours des deux dernières décennies, soit le coronavirus du syndrome respiratoire aigu sévère 1 (SRAS-CoV-1), le coronavirus du syndrome respiratoire du Moyen-Orient (SRMO-CoV) et le coronavirus du syndrome respiratoire aigu sévère 2 (SRAS-CoV-2). Le SRAS-CoV-2 cause la COVID-19, qui provoque la morbidité répandue observée dans le cadre de la pandémie actuelle.
La structure des coronavirus est relativement simple : ils comportent, en leur centre, un seul brin d’ARN, entouré d’une capside et d’une enveloppe externe. Le virus est constitué de quatre protéines structurales, soit la nucléocapside (N), la membrane (M), l’enveloppe (E) et la protéine de spicule (S)7. La protéine S forme des trimères qui semblent jaillir de l’enveloppe virale, donnant l’impression d’une couronne, d’où le nom de « coronavirus ». La protéine de spicule joue un rôle crucial dans l’infection, car elle contient le domaine de liaison aux récepteurs qui permet la fixation du virus à la cellule hôte. Les protéines S du SRAS-CoV-1 et du SRAS-CoV-2, qui sont étroitement liés, se fixent à une protéine courante présente à la surface des cellules, l’enzyme de conversion de l’angiotensine 2 (ACE2, pour angiotensin converting enzyme 2), pour pénétrer dans les cellules hôtes8,9. L’ACE2 est souvent exprimée dans les tissus cardiopulmonaires (du cœur et de l’appareil respiratoire), le tractus gastro-intestinal, les cellules immunitaires (monocytes, macrophages et cellules dendritiques) et d’autres tissus; sa principale fonction est de réguler la tension artérielle10. La protéine S est la protéine immunodominante, c’est-à-dire qu’elle déclenche de puissantes réponses des lymphocytes T et des anticorps, et qu’elle est une cible attrayante pour les vaccins et les thérapies, en raison de son rôle dans la fixation et la pénétration des virus. Toutefois, les lymphocytes T et B sont également capables de cibler les protéines M et N11.
Bien que peu d’études aient examiné l’immunité innée au SRAS-CoV-2, il semble bien que, comme dans le cas d’autres coronavirus, celui-ci déclenche une variété de réponses immunitaires innées, faisant interagir notamment les interférons (IFN) de types I et III, qui sont les principaux facteurs antiviraux produits par les cellules, ainsi que des cytokines pro-inflammatoires, comme le facteur de nécrose tumorale-alpha (TNF-⍺) et les interleukines (IL) 1 et 612. Les premières études indiquent que le SRAS-CoV-2 inhibe les IFN de types I et III dans une plus grande mesure que le fait le SRAS-CoV-113. Cependant, il provoque une puissante réponse pro-inflammatoire des cytokines14, et ce déséquilibre entre une faible réponse antivirale innée et une importante inflammation pourrait contribuer à l’apparition de la nouvelle maladie à coronavirus de 2019 (COVID-19). En effet, l’antagonisme de l’IFN a été corrélé à une gravité accrue de la COVID-1915.
Des renseignements sur les réponses immunitaires acquises au SRAS-CoV-2 commencent à émerger. Bien que des réponses des lymphocytes T aux protéines S et N du virus aient été observées dans la plupart des études cliniques, les patients atteints de formes plus graves de la maladie semblent présenter une lymphopénie importante caractérisée par une forte chute du nombre de lymphocytes T dans le sang16,17. Chez les patients atteints d’une forme grave de la COVID-19, les lymphocytes T sont activés, mais présentent essentiellement une réponse inflammatoire ainsi que les caractéristiques de l’épuisement18, alors que les personnes atteintes d’une forme légère ont des lymphocytes T plus efficaces et multifonctionnels, dont des lymphocytes T CD8 capables de tuer les cellules infectées par le virus et des lymphocytes T CD4 produisant une variété de cytokines qui participent à d’autres aspects de la réponse immunitaire19,20. En général, les réponses des lymphocytes T au virus tendent vers l’inflammation, ce qui peut contribuer à la gravité de la COVID-19. D’autres études ont révélé que, chez les patients gravement malades, alors que le nombre de lymphocytes T est réduit dans le sang, un grand nombre s’accumule dans les poumons et contribue probablement au syndrome de détresse respiratoire aiguë (SDRA)21,22 en provoquant un choc cytokinique caractérisé par des taux incontrôlés de cytokines inflammatoires (syndrome de relargage de cytokines)23 (explications à la section 7).
La réponse humorale au SRAS-CoV-2 est vastement étudiée, étant donné l’importance attendue des anticorps dans la protection contre une réinfection. Plusieurs études ont montré qu’une infection par le SRAS-CoV-2 induit une solide réponse des anticorps dans les 7 à 14 jours, des anticorps IgM, IgG et IgA ayant été détectés chez presque toutes les personnes infectées, les IgG persistant durant les semaines suivant le rétablissement24. Les anticorps les plus fréquemment détectés sont dirigés contre les protéines N et S du SRAS-CoV-225,26. Les anticorps dirigés contre le domaine de liaison aux récepteurs de la protéine S présentent un intérêt, car ils peuvent empêcher le virus de se fixer au récepteur d’entrée, l’ACE2, sur les cellules hôtes. Les anticorps spécifiques au domaine de liaison aux récepteurs sont des anticorps neutralisants très efficaces et ils ont été décelés chez la plupart des patients27,28. On ignore encore la durée de la réponse des anticorps; reste aussi à savoir si toutes les personnes infectées produisent de grandes quantités de ces anticorps neutralisants et si ces derniers sont en corrélation avec la protection. Des études portant sur d’autres infections par des coronavirus révèlent que des infections moins graves et une maladie légère provoquent une production d’anticorps de courte durée29,30, alors que le SRAS‑CoV‑1 et le SRMO-CoV, qui causent des maladies plus graves, entraînent des réponses plus longues, qui durent de deux à trois ans29,31,32, certains rapports révélant une réponse immunitaire secondaire encore plus longue33.
3. Évaluation de la réponse immunitaire ou dépistage du virus
Étant donné que le SRAS-CoV-2 est un nouveau coronavirus qui s’est propagé à une vitesse sans précédent à travers le monde, la mise au point d’épreuves diagnostiques a été une réelle course contre la montre. Dès les premières semaines de la propagation de l’infection à Wuhan, il est devenu évident que la lutte contre la dissémination de ce nouveau virus serait très difficile, en raison de sa grande infectiosité, du fait que les personnes atteintes ne présentaient pas toutes la maladie caractérisée, et que le virus peut être transmis par des personnes ne présentant que de légers symptômes, voire aucun symptôme. Au cours des derniers mois, les données épidémiologiques ont révélé que le nombre moyen de personnes infectées par une personne si aucune mesure de contrôle n’est en place (le taux de reproduction de base, ou « R0 ») du SRAS-CoV-2 est autour de 2,2 à 2,634–37. Cela signifie que ce virus est plus infectieux que celui de la grippe (R0 de 1,838), mais moins que celui de la rougeole (R0 de 1539). Bien que le taux de transmission asymptomatique varie considérablement selon la région géographique et les mesures de santé publique en place dans les différents pays, certaines estimations indiquent que la transmission asymptomatique contribue jusqu’à hauteur de 40 à 45 % du nombre total de cas40.
Analyses moléculaires : Dès les premières semaines de l’émergence de la COVID-19, les premières épreuves diagnostiques mises au point étaient des analyses moléculaires par amplification des acides nucléiques. Il s’agit de tests fondés sur une réaction en chaîne de la polymérase (PCR, pour polymerase chain reaction) qui permettent de détecter le matériel génétique viral isolé d’échantillons prélevés dans la gorge ou le nez. On peut aussi utiliser des échantillons d’expectorations ou de lavages broncho-alvéolaires si l’infection s’est propagée jusqu’aux poumons41. Le procédé de base consiste à isoler l’ARN viral des échantillons biologiques, à le convertir en ADN au moyen de la transcriptase inverse, une enzyme, puis à amplifier l’ADN par PCR, à l’aide d’amorces spécifiques au SRAS-CoV-2. Cette méthode, qui a rapidement été diffusée et publiée, est maintenant largement utilisée partout dans le monde42,43. Bien que ce type d’épreuve soit exigeant en main-d’œuvre, il peut détecter une infection active avec une haute sensibilité44. En l’absence de vaccins et de traitements, cette épreuve a joué un rôle crucial dans la stratégie mondiale de santé publique pour contenir les infections par le SRAS-CoV-2, grâce à un dépistage, un repérage et une mise en isolement rapide des cas. La limite de cette épreuve réside dans le fait qu’elle sert généralement au diagnostic des infections actives chez les personnes symptomatiques. Étant donné que le virus peut être transmis par des personnes asymptomatiques, il est difficile de retracer tous les cas infectés au moyen de ce test.
Analyses sérologiques : Ce type d’épreuves détecte la présence d’anticorps contre le SRAS-CoV-2 dans le sang45. Les anticorps apparaissent presque universellement à la suite d’une infection par le virus, chez toutes les personnes qui ne sont pas immunodéprimées, d’une à deux semaines après l’infection24. Ainsi, l’analyse sérologique peut non seulement confirmer la présence d’une infection virale chez les personnes qui présentent des symptômes, mais aussi chez celles dont l’infection est asymptomatique. Comme les anticorps sont probablement présents dans le sang pendant des semaines, voire des mois, après l’infection, ces analyses peuvent être effectuées longtemps après le rétablissement des personnes, afin de repérer celles qui ont déjà été infectées par le SRAS-CoV-2. Toutefois, il y a un délai entre le début de l’infection et l’apparition des anticorps dans le sang; par conséquent, les analyses sérologiques ne sont pas une façon fiable d’établir un diagnostic de l’infection active. De nombreuses analyses sérologiques commercialisées sont basées sur le principe d’immunochromatographie à flux latéral, méthode qui consiste à laisser couler le liquide contenant les anticorps (le sang du patient) sur un substrat solide présentant des antigènes du SRAS-CoV-2. Si des anticorps sont présents dans le sang, ils se lieront aux protéines virales, ce qui causera un changement de couleur indiquant un résultat positif. Ce type d’analyse fondée sur une détection simple positive ou négative d’anticorps est utile pour la surveillance à grande échelle, mais ne fournit pas de renseignements concernant la quantité, le type ou la fonction des anticorps. La méthode ELISA, un essai d’immuno-absorption enzymatique, convient mieux à la détection précise d’anticorps; il s’agit d’une épreuve de laboratoire courante qui peut mesurer non seulement la présence, mais aussi le titre (quantité) et le type (IgG, IgM, IgA) d’anticorps. Cette méthode permet de mieux mesurer la puissance de la réponse humorale46. En général, plus le titre des anticorps est élevé, meilleure est la protection. La méthode ELISA est complexe et ne peut être réalisée qu’en laboratoire, alors que les analyses à flux latéral sont présentées comme étant peu coûteuses et faciles à réaliser au point de traitement. Toutefois, de nombreuses analyses à flux latéral sont en cours d’examen afin d’évaluer plus rigoureusement leur sensibilité (fréquence à laquelle elles ne détectent pas un échantillon positif) et leur spécificité (fréquence à laquelle le résultat est positif, alors que l’échantillon est en réalité négatif), deux caractéristiques qui ont d’importantes répercussions sur la santé publique. Si l’on confirme que la présence d’anticorps protège réellement contre une réinfection, la réalisation d’analyses sérologiques dans de vastes populations (sérosurveillance) peut jouer un rôle important dans l’évaluation du taux d’immunisation de la population (immunité collective) et la validation du concept de « certificat d’immunité »47.
Méthode de neutralisation virale : La seule présence d’anticorps dans le sang, mesurée par analyses à flux latéral ou par la méthode ELISA, ne démontre pas la capacité de ces anticorps à bloquer efficacement une infection virale. La référence absolue pour la mesure de la capacité d’un anticorps à bloquer (ou « neutraliser ») un virus est le test de séroneutralisation par réduction des plages de lyse48. Lorsque des virus se répliquent à l’intérieur de cellules mises en culture, ils sont ensuite libérés lorsque les cellules sont tuées (ce qu’on appelle la « lyse »). Ce phénomène crée des trous, qu’on nomme « plages », qui peuvent facilement être comptés sous microscope ou quantifiés selon des paramètres automatisés. Si les anticorps inhibent la capacité du virus à infecter les cellules, peu ou pas de plages se formeront après qu’un mélange des anticorps et du virus aura été étendu sur les cellules, ce qui indique que les anticorps peuvent « neutraliser » le virus. Cette méthode mesure non seulement le titre des anticorps, mais aussi la capacité de ces derniers à protéger l’organisme contre l’infection virale. Toutefois, cette méthode requiert beaucoup de main-d’œuvre et, comme elle nécessite l’utilisation du virus infectieux vivant, elle doit être réalisée dans des laboratoires pouvant assurer les conditions de biosécurité de niveau 348. D’autres analyses reposant sur des virus substituts (méthodes de neutralisation de pseudovirus) ont été mises au point, mais ne sont actuellement utilisées que dans des laboratoires de recherche.
En résumé, un certain nombre d’épreuves ont été rapidement mises au point et adaptées à partir des méthodes existantes afin de fournir diverses façons de dépister une infection par le SRAS-CoV-2 ainsi que d’évaluer la réponse des anticorps de l’hôte. Les épreuves diagnostiques qui détectent la présence du virus sont complémentaires aux méthodes qui mesurent l’immunité de l’hôte, car ensemble, elles peuvent diagnostiquer les infections actives et asymptomatiques. Bien que ces méthodes ne soient pas toutes complètement optimisées pour le moment, elles ont joué un rôle essentiel dans la stratégie actuelle de santé publique visant à dépister, repérer et isoler les cas, permettant ainsi de sauver des millions de vies partout dans le monde.
4. Réponse des anticorps au SRAS-CoV-2 et séroprévalence dans la population
Alors que la plupart des études examinant les réponses des anticorps ont révélé que presque toutes les personnes infectées par le SRAS-CoV-2 produisent des anticorps contre le virus, on ignore encore la durée de cette production et la capacité de ces anticorps à conférer une protection contre une réinfection. Bien que certaines personnes produisent des anticorps dès huit jours après l’infection, dans la plupart des études, cette production atteint son maximum après environ 14 jours, et on observe une variation importante d’un patient à l’autre24. Les IgM ont été les premiers anticorps détectés au cours de la première semaine, mais leur taux diminue après 30 jours, alors que le taux des IgG augmente lentement et demeure élevé jusqu’à 25 semaines après l’infection. Les titres des anticorps sont en corrélation avec la gravité de l’infection, les taux d’anticorps étant plus élevés chez les personnes ayant été aux prises avec de plus graves symptômes. La majorité des anticorps détectés était dirigée contre les protéines N et S du SRAS‑CoV‑2. Plus particulièrement, les anticorps dirigés contre le domaine de liaison au récepteur de la protéine S, qui est hautement immunogène, ont le potentiel d’être hautement neutralisants27,28. Les études ont révélé que, durant la phase précoce de la réponse immunitaire, le titre des anticorps est en corrélation avec la neutralisation, les anticorps neutralisants diminuant rapidement à la suite du rétablissement49. Des études précédentes ont révélé que les coronavirus qui causent des infections plus graves, comme le SRAS-CoV-1 et le SRMO-CoV, induisent une production d’anticorps plus importante et plus durable, comparativement aux coronavirus qui causent des infections plus légères29,31,32. Jusqu’à maintenant, on signale peu de cas de réinfection chez les personnes qui s’étaient rétablies de la COVID‑19, mais des études longitudinales doivent être réalisées afin de connaître la durée de la protection et de savoir si les personnes ayant présenté des symptômes légers disposeront d’une protection d’une plus courte durée que celles ayant subi une forme plus grave de la maladie.
On estime que de 60 à 70 % de la population devraient avoir produit des anticorps avant qu’une immunité collective contre le SRAS-CoV-2 puisse exister50. Des enquêtes séroépidémiologiques menées dans des pays membres de l’Union européenne et de l’Espace économique européen révèlent un faible taux de séropositivité, allant d’environ 1 % en Finlande51 et en Écosse52, à 5 % en Espagne53 et 8,5 % au Royaume-Uni54. Ces résultats sont en phase avec ceux des études de séroprévalence réalisées dans d’autres pays55–57 qui indiquent que, dans l’ensemble, moins de 10 % de la population de la plupart des pays ont été exposés au virus. Des travaux sont en cours à l’échelle mondiale afin de déterminer la séroprévalence précise des anticorps contre le SRAS-CoV-2. Cependant, les estimations actuelles conduisent à penser qu’il est très peu probable qu’une immunité collective sera atteinte d’ici peu, dans quelque pays que ce soit à travers le monde.
5. Anticorps en tant que traitements
Les anticorps neutralisants qui bloquent l’entrée du virus ont le potentiel d’être utilisés tant comme traitement qu’en prophylaxie. Le transfert de plasma de convalescence, qui implique la transfusion de l’élément liquide du sang de personnes qui se sont rétablies d’une infection à des personnes infectées, a été essayé comme traitement salvateur pour de nombreuses maladies infectieuses, avec divers degrés de réussite. Le plasma des personnes rétablies contient divers anticorps, y compris des anticorps neutralisants, dirigés contre le pathogène, et peut empêcher l’infection de nouvelles cellules chez la personne qui reçoit ce plasma et lui permettre de se rétablir; toutefois, l’immunité conférée par ce type de traitement est de courte durée. Les quelques études ayant évalué l’efficacité des transfusions de plasma de convalescence à des personnes atteintes d’une forme grave de la COVID-19 ont produit des résultats variés58–62, probablement parce que le succès de cette approche repose sur la présence de titres élevés d’anticorps neutralisants ainsi que sur le moment de l’administration du traitement. On doit aussi tenir compte d’autres éléments si l’on choisit les transfusions de plasma de convalescence, notamment le dépistage d’autres agents infectieux, l’appariement du groupe sanguin, ainsi que les préoccupations potentielles concernant la présence d’anticorps qui pourraient exacerber la maladie en raison de leur capacité à provoquer une inflammation par réaction dépendant des anticorps (antibody dependent enhancement). L’administration d’anticorps monoclonaux (dérivés d’une unique cellule plasmatique qui produit un type d’anticorps ainsi qu’une reproductibilité et une efficacité supérieures en matière d’activité biologique) est une solution de rechange à l’utilisation de plasma de convalescence, car elle contourne la plupart de ces limites63,64. D’importants progrès ont été réalisés afin de repérer et de produire rapidement les anticorps monoclonaux pouvant neutraliser des virus. Dans le cadre des récentes approches, des cellules de patients rétablis qui produisent un anticorps unique sont isolées et immortalisées; on intensifie ensuite rapidement la production des anticorps et les essais. Cette méthode a permis de mettre au point le mAb114, constitué d’un seul anticorps, et le RGEN-EB3, qui contient un cocktail de trois anticorps, pour lutter contre le virus Ebola. Ces deux traitements se sont avérés efficaces pour réduire la mortalité dans le cadre d’essais randomisés65. Plusieurs rapports font état d’anticorps monoclonaux isolés chez des donneurs convalescents de la COVID-19 ayant réduit la charge virale du domaine de liaison aux récepteurs dans les poumons de modèles animaux66,67. De tels traitements, ainsi que d’autres fondés sur les anticorps monoclonaux, devraient bientôt faire l’objet d’essais cliniques et sont parmi les traitements les plus prometteurs contre la COVID-19 qui ont le potentiel d’être hautement efficaces et peuvent être rapidement produits à grande échelle.
6. Corrélats de protection contre la COVID-19
Le terme « corrélat de protection » a d’abord été utilisé dans le contexte de la vaccination afin de parler de la réponse immunitaire responsable de la protection et statistiquement interreliée avec cette dernière68. Étant donné que la plupart des vaccins courants induisent la production d’anticorps sériques, ce terme désigne généralement le taux d’anticorps produits après la vaccination qui confère la protection contre l’infection. Pour de nombreux vaccins, ces taux ont été établis de façon empirique, soit en provoquant le système immunitaire des personnes vaccinées en leur administrant le pathogène soit par de vastes études menées à la suite de la vaccination afin de déterminer le titre d’anticorps qui confère une protection. Par exemple, des taux d’anticorps sériques de 0,01 à 0,1 UI/ml et de 0,5 UI/ml sont considérés comme des corrélats de protection contre la diphtérie69 et la rage70, respectivement. En ce qui concerne le vaccin contre la grippe saisonnière, un titre d’anticorps de 1:40 au test par inhibition de l’hémagglutinine est considéré comme protecteur71. Dans le cas de vaccins contre certains pathogènes comme le virus varicelle-zona et Mycobacterium tuberculosis, les corrélats de protection connus concernent les lymphocytes T CD4+72 et l’interféron73, respectivement, mais les taux qui confèrent une protection n’ont pas été déduits.
Outre les vaccins, les infections naturelles confèrent également une protection contre les expositions subséquentes; par conséquent, les corrélats de protection sont aussi pertinents dans ce contexte. Dans le cadre de la pandémie actuelle de COVID-19, la notion du certificat d’immunité est un sujet fréquent de discussion; ce concept repose sur l’hypothèse qu’en augmentant le dépistage des anticorps, il serait possible de repérer les personnes immunisées contre la COVID-19, ce qui permettrait d’estimer l’immunité collective et de connaître les personnes qui peuvent réintégrer la population active sans craindre d’être infectées47. Toutefois, cette discussion est prématurée, car on ignore actuellement quels sont les corrélats de protection contre la COVID-19, bien que des études portant sur les taux et les fonctions des anticorps commencent à fournir certains renseignements74. Bien que presque toutes les personnes infectées par le SRAS-CoV-2 développent des anticorps, la majorité d’entre elles (de 80 à 90 %) présente des taux globaux d’anticorps et des titres d’anticorps neutralisants faibles49. La production d’anticorps semble aussi être corrélée à la gravité de la maladie, ce qui implique que, chez les personnes ayant eu une infection de faible intensité, la réponse immunitaire peut aussi avoir été faible, et la production d’anticorps pourrait ne pas être assez puissante pour conférer une protection29,30. En outre, des études récentes ont révélé que la plupart des personnes qui présentent la maladie produisent des anticorps neutralisants, mais que le taux de ces derniers commence à décroître à l’intérieur de huit semaines49,75.
La contribution des lymphocytes T à la protection contre une réinfection par le virus de la COVID-19 est encore inconnue, et il est possible qu’ils contribuent aussi à une telle protection. Les coronavirus soulèvent aussi une préoccupation concernant le fait qu’un grand nombre d’entre eux produisent des réponses immunitaires inflammatoires pouvant exacerber la maladie, les lymphocytes T et les anticorps pouvant contribuer à ce phénomène12. Par conséquent, il sera nécessaire de mener des études plus approfondies pour mesurer la réponse immunitaire des personnes présentant tous les degrés de gravité de la maladie. Les données préliminaires révèlent que les macaques rhésus réexposés au SRAS-CoV-2 après une primo-infection étaient protégés. Il s’agit d’un résultat prometteur, car la protection était en corrélée à une importante production d’anticorps neutralisants chez les singes74. Il sera nécessaire de réaliser des études longitudinales afin de faire le suivi des personnes qui se sont rétablies de la COVID-19, pour surveiller les cas de réinfection, et d’établir une corrélation entre les taux d’anticorps et les réinfections. Ces études permettront de déterminer si, en effet, les anticorps suffisent et, dans l’affirmative, quel taux est nécessaire pour conférer une protection.
7. Effets délétères de la réponse immunitaire aux coronavirus
Bien que les réponses immunitaires aux virus jouent un rôle essentiel dans l’élimination des infections et dans la protection contre les réinfections, la réponse immunitaire pro-inflammatoire peut aussi être associée à l’issue et à la gravité de la maladie, particulièrement à la suite d’infections virales respiratoires. Le virus respiratoire syncytial (VRS), le virus de la grippe et l’adénovirus peuvent tous provoquer une pneumonie associée à une réponse immunitaire pro-inflammatoire de l’hôte76. Le SRAS-CoV-1 et le SRMO-CoV ont aussi été associés à des réponses immunitaires excessives qui peuvent provoquer une forme grave de la maladie caractérisée par des symptômes de fièvre et de pneumonie, suivis par le syndrome de détresse respiratoire aiguë (SDRA), qui serait le résultat d’un syndrome de relargage de cytokines (SRC)77. Environ 20 % des patients infectés par le SRAS-CoV-2 viennent à présenter des symptômes graves, et environ 5 % présentent une détresse respiratoire aiguë14,78–81. Chez les patients aux prises avec des maladies concomitantes, telle une maladie cardiovasculaire, la morbidité est plus élevée82. Comme c’est le cas des autres coronavirus, la forme grave de la maladie causée par le SRAS-CoV-2 apparaît de sept à dix jours après la manifestation des symptômes, ce qui correspond au début de la réponse excessive incontrôlée de l’hôte, plutôt qu’à un effet direct de la virémie83. Les symptômes associés à la détresse respiratoire aiguë causée par le SRAS-CoV-2 sont semblables à ceux du SRC, mais les mécanismes sous‑jacents menant à cette morbidité sont encore à l’étude. Nous savons maintenant que le SRAS-CoV-2 infecte diverses cellules de l’hôte, notamment celles des tissus des voies respiratoires et cardiovasculaires, ainsi que les cellules immunitaires, y compris les monocytes et les macrophages et, possiblement, les cellules dendritiques. Le sérum des patients atteints de la forme grave de la maladie présente des taux élevés de protéine C réactive, d’IL-6 et d’autres cytokines inflammatoires, qui sont toutes associées à des issues cliniques défavorables84,85. Une lymphopénie caractérisée par une diminution des taux de CD4, de CD8 et de cellules NK est en corrélation avec la gravité de la COVID-1986. On a aussi signalé la présence de facteurs des lymphocytes T évoquant un épuisement et de monocytes inflammatoires18.
En plus de l’inflammation associée aux lymphocytes T, on se préoccupe du fait que la production d’anticorps contre le SRAS-CoV-2 puisse contribuer à la pathologie respiratoire, en raison d’une réaction dépendant des anticorps. D’autres coronavirus, comme le SRAS-CoV-1, ont été associés à une telle réaction caractérisée par une corrélation entre des titres d’anticorps plus élevés et une morbidité plus grave, voire le décès87,88. La réaction dépendant des anticorps est un phénomène associé à la présence d’anticorps IgG non neutralisants qui se lient au virus, puis à des cellules qui expriment un récepteur des anticorps spécifique (récepteur Fc). Ces liaisons provoquent l’activation de certaines cellules qui expriment le récepteur Fc, comme les monocytes et les macrophages, ainsi que la production de cytokines inflammatoires et l’accumulation de ces cellules dans les poumons, ce qui cause des lésions89. Des études sont en cours afin de déterminer si la réaction dépendant des anticorps est observée chez les patients présentant des formes graves de la COVID-19.
En résumé, les patients aux prises avec une forme grave de la COVID-19 présentent une détresse respiratoire aiguë associée au SRC, possiblement causé par une inflammation incontrôlée. La cause sous-jacente pourrait être l’activation de certaines cellules immunitaires, y compris les monocytes, les macrophages, les cellules dendritiques et les lymphocytes T, et l’absence ou l’incapacité des mécanismes qui, normalement, régulent la réponse immunitaire. Les récentes études qui montrent les bienfaits de l’utilisation de la dexaméthasone, un corticostéroïde immunosuppresseur, chez des patients aux prises avec une forme grave de la maladie corroborent la conclusion qu’une inflammation incontrôlée est à la base de la morbidité associée à la COVID-1990.
Bibliographie
1.
WOO, P. C. Y., S. K. P. LAU, Y. HUANG, et K.-Y. YUEN, « Coronavirus Diversity, Phylogeny and Interspecies Jumping », Exp. Biol. Med., vol. 234, 2009, p. 1117–1127.
2.
BRACIALE, T. J., et Y. S. HAHN, « Immunity to viruses », Immunological Reviews, vol. 255, 2013, p. 5–12.
3.
DUAN, L., et E. MUKHERJEE, « Focus: Microbiome », Janeway’s Immunobiology, Ninth Edition, Yale J. Biol. Med., vol. 89, 2016, 424 p.
4.
CUI, J., F. LI, et Z. L. SHI, « Origin and evolution of pathogenic coronaviruses », Nature Reviews Microbiology, vol. 17, 2019, p. 181–192.
5.
GORBALENYA, A. E. et collab., « The species Severe acute respiratory syndrome-related coronavirus: classifying 2019-nCoV and naming it SARS-CoV-2 », Nature Microbiology, vol. 5, 2020, p. 536–544.
6.
TANG, X. et collab., « On the origin and continuing evolution of SARS-CoV-2 », Natl. Sci. Rev., 2020, nwaa036, doi : 10.1093/nsr.
7.
JIANG, S., C. HILLYER et L. DU, « Neutralizing Antibodies against SARS-CoV-2 and Other Human Coronaviruses », Trends in Immunology, vol. 41, 2020, p. 355–359.
8.
LU, R. et collab., « Genomic characterisation and epidemiology of 2019 novel coronavirus: implications for virus origins and receptor binding », Lancet, vol. 395, 2020, p. 565–574.
9.
WAN, Y., J. SHANG, R. GRAHAM, R. S. BARIC et F. LI, « Receptor Recognition by the Novel Coronavirus from Wuhan: an Analysis Based on Decade-Long Structural Studies of SARS Coronavirus », J. Virol, 2020, 94 p.
10.
DU, M. et collab., « Multiomics Evaluation of Gastrointestinal and Other Clinical Characteristics of COVID19 », Gastroenterology, vol. 158, 2020, p. 2298‑2301.e7.
11.
GRIFONI, A. et collab., « A Sequence Homology and Bioinformatic Approach Can Predict Candidate Targets for Immune Responses to SARS-CoV-2 », Cell Host Microbe, vol. 27, 2020, p. 671-680.e2.
12.
VABRET, N. et collab., « Immunology of COVID-19: Current State of the Science », Immunity, vol. 52, 2020, p. 910–941.
13.
BLANCO-MELO, D. et collab., « Imbalanced Host Response to SARS-CoV-2 Drives Development of COVID19 », Cell, vol. 181, 2020.
14.
HUANG, C. et collab., « Clinical features of patients infected with 2019 novel coronavirus in Wuhan, China », Lancet, vol. 395, 2020, p. 497–506.
15.
HADJADJ, J. et collab., « Impaired type I interferon activity and exacerbated inflammatory responses in severe Covid-19 patients », medRxiv, 2020, doi :10.1101/2020.04.19.20068015.
16.
WANG, F. et collab., « Characteristics of Peripheral Lymphocyte Subset Alteration in COVID-19 Pneumonia », J. Infect. Dis., vol. 221, 2020, p. 1762–1769.
17.
ZHENG, M. et collab., « Functional exhaustion of antiviral lymphocytes in COVID-19 patients », Cellular and Molecular Immunology, vol. 17, 2020, p. 533–535.
18.
DIAO, B. et collab., « Reduction and Functional Exhaustion of T Cells in Patients With Coronavirus Disease 2019 (COVID-19) », Front. Immunol., vol. 11, 2020, 827 p.
19.
LIU, B. et collab., « Persistent SARS-CoV-2 presence is companied with defects in adaptive immune system in non-severe COVID-19 patients », medRxiv, 2020, http://medrxiv.org/lookup/doi/10.1101/2020.03.26.20044768, doi : 10.1101/2020.03.26.20044768.
20.
THEVARAJAN, I. et collab., « Breadth of concomitant immune responses prior to patient recovery: a case report of non-severe COVID-19 », Nature Medicine, vol. 26, 2020, p. 453–455.
21.
ZHOU, Y. et collab., « Pathogenic T cells and inflammatory monocytes incite inflammatory storm in severe COVID-19 patients », Natl. Sci. Rev., 2020, nwaa041.
22.
XU, Z. et collab., « Pathological findings of COVID-19 associated with acute respiratory distress syndrome », Lancet Respir. Med., vol. 8, 2020, p. 420–422. 23. MOORE, J. B. et C. H. JUNE, « Cytokine release syndrome in severe COVID-19 », Science, vol. 368, 2020, p. 473–474.
23.
HUANG, A. T. et collab., « A systematic review of antibody mediated immunity to coronaviruses: antibody kinetics, correlates of protection, and association of antibody responses with severity of disease », medRxiv, 2020, doi : 10.1101/2020.04.14.20065771.
24.
AMANAT, F. et collab., « A serological assay to detect SARS-CoV-2 seroconversion in humans », Nat. Med., 2020, p. 1–4, doi : 10.1038/s41591-020-0913-5.
25.
TO, K. K. W. et collab., « Temporal profiles of viral load in posterior oropharyngeal saliva samples and serum antibody responses during infection by SARS-CoV-2: an observational cohort study », Lancet Infect. Dis., vol. 20, 2020, p. 565–574.
26.
JU, B. et collab., « Human neutralizing antibodies elicited by SARS-CoV-2 infection », Nature, 2020, p. 1–8, doi : 10.1038/s41586-020-2380-z.
27.
WU, F. et collab., « Neutralizing antibody responses to SARS-CoV-2 in a COVID-19 recovered patient cohort and their implications », medRxiv, 2020.
28.
CAO, W. C., W. LIU, P. H. ZHANG, F. ZHANG et J. H. RICHARDUS, « Disappearance of antibodies to SARS-associated coronavirus after recovery », New England Journal of Medicine, vol. 357, 2007, p. 1162–1163.
29.
CALLOW, K. A., H. F. PARRY, M. SERGEANT et D. A. J. TYRRELL, « The time course of the immune response to experimental coronavirus infection of man », Epidemiol. Infect., vol. 105, 1990, p. 435–446.
30.
WU, L. P. et collab., « Duration of antibody responses after severe acute respiratory syndrome », Emerg. Infect. Dis., vol. 13, 2007, p. 1562–1564.
31.
KO, J. H. et collab., « Serologic responses of 42 MERS-coronavirus-infected patients according to the disease severity », Diagn. Microbiol. Infect. Dis., vol. 89, 2017, p. 106–111.
32.
PAYNE, D. C. et collab., « Persistence of antibodies against middle east respiratory syndrome coronavirus », Emerg. Infect. Dis., vol. 22, 2016, p. 1824–1826.
33.
LI, Q. et collab., « Early Transmission Dynamics in Wuhan, China, of Novel Coronavirus–Infected Pneumonia », N. Engl. J. Med., vol. 382, 2020, p. 1199–1207.
34.
ALIMOHAMADI, Y., M. TAGHDIR et M. SEPANDI, « Estimate of the Basic Reproduction Number for COVID-19: A Systematic Review and Meta-analysis », J. Prev. Med. Public Heal., vol. 53, 2020, p. 151–157.
35.
“When will it be over?”: An introduction to viral reproduction numbers, R0 and Re, CEBM, https://www.cebm.net/covid-19/when-will-it-be-over-an-introduction-to-viral-reproduction-numbers-r0-and-re/.
36.
D’ARIENZO, M. et A. CONIGLIO, « Assessment of the SARS-CoV-2 basic reproduction number, R0, based on the early phase of COVID-19 outbreak in Italy », Biosaf. Heal., 2020, doi :10.1016/j.bsheal.2020.03.004.
37.
BIGGERSTAFF, M., S. CAUCHEMEZ, C. REED, M. GAMBHIR et L. FINELLI, « Estimates of the reproduction number for seasonal, pandemic, and zoonotic influenza: a systematic review of the literature », BMC Infect. Dis., vol. 14, 2014, p. 480.
38.
GUERRA, F. M. et collab., « The basic reproduction number (R0) of measles: a systematic review », The Lancet Infectious Diseases, vol. 17, 2017, e420–e428.
39.
ORAN, D. P. et E. J. TOPOL, « Prevalence of Asymptomatic SARS-CoV-2 Infection », Ann. Intern. Med., 2020, doi : 10.7326/m20-3012.
40.
LOEFFELHOLZ, M. J. et Y. W. TANG, « Laboratory diagnosis of emerging human coronavirus infections– the state of the art », Emerging Microbes and Infections, vol. 9, 2020, p. 747–756.
41.
CORMAN, V. M. et collab., « Detection of 2019 novel coronavirus (2019-nCoV) by real-time RT-PCR », Eurosurveillance, vol. 25, 2020.
42.
DHAMA, K. et collab., « Coronavirus Disease 2019–COVID-19 », Clin. Microbiol. Rev., vol. 33, 2020.
43.
WÖLFEL, R. et collab., « Virological assessment of hospitalized patients with COVID-2019 », Nature, vol. 581, 2020, p. 465– 469.
44.
Serology Testing for COVID-19 at CDC, CDC, https://www.cdc.gov/coronavirus/2019-ncov/lab/serology-testing.html.
45.
XIAO, S., Y. WU et H. LIU, « Evolving status of the 2019 novel coronavirus infection: Proposal of conventional serologic assays for disease diagnosis and infection monitoring », J. Med. Virol, vol. 92, 2020, p. 464–467.
46.
‘Immunity passports’ in the context of COVID-19, https://www.who.int/news-room/commentaries/detail/immunity-passports-in-the-context-of-covid-19.
47.
KRAMMER, F. et V. SIMON, « Serology assays to manage COVID-19 », Science, vol. 368, 2020, p. 1060–1061.
48.
LONG, Q.-X. et collab., « Clinical and immunological assessment of asymptomatic SARS-CoV-2 infections », Nat. Med., 2020, p. 1–5, doi :10.1038/s41591-020-0965-6.
49.
GOMES, M. G. M. et collab., « Individual variation in susceptibility or exposure to SARS-CoV-2 lowers the herd immunity threshold », medRxiv, 2020, http://medrxiv.org/lookup/doi/10.1101/2020.04.27.20081893, doi : 10.1101/2020.04.27.20081893.
50.
HAVERI, A. et collab., « Serological and molecular findings during SARS-CoV-2 infection: The first case study in Finland, January to February 2020 », Eurosurveillance, vol. 25, 2020.
51.
THOMPSON, C. et collab., « Neutralising antibodies to SARS coronavirus 2 in Scottish blood donors - a pilot study of the value of serology to determine population exposure », medRxiv, 2020, http://medrxiv.org/lookup/doi/10.1101/2020.04.13.20060467 doi : 10.1101/2020.04.13.20060467.
52.
Immune responses and immunity to SARS-CoV-2, https://www.ecdc.europa.eu/en/covid-19/latest-evidence/immune-responses.
53.
Sero-surveillance of COVID-19, GOV.UK., https://www.gov.uk/government/publications/nationalcovid-19-surveillance-reports/sero-surveillance-of-covid-19.
54.
STRINGHINI, S. et collab., « Seroprevalence of anti-SARS-CoV-2 IgG antibodies in Geneva, Switzerland (SEROCoV-POP): a population-based study », Lancet, vol. 0, 2020.
55.
XU, X. et collab., « Seroprevalence of immunoglobulin M and G antibodies against SARS-CoV-2 in China », Nat. Med., 2020, p. 1–3, doi : 10.1038/s41591-020-0949-6.
56.
SOOD, N. et collab., « Seroprevalence of SARS-CoV-2–Specific Antibodies Among Adults in Los Angeles County, California, on April 10-11, 2020 », JAMA, vol. 323, 2020, 2425 p.
57.
AHN, J. Y. et collab., « Use of convalescent plasma therapy in two covid-19 patients with acute respiratory distress syndrome in Korea », J. Korean Med. Sci., vol. 35, 2020.
58.
DUAN, K. et collab., « Effectiveness of convalescent plasma therapy in severe COVID-19 patients », Proc. Natl. Acad. Sci. U. S. A., vol. 117, 2020, p. 9490–9496.
59.
SHEN, C. et collab., « Treatment of 5 Critically Ill Patients with COVID-19 with Convalescent Plasma », JAMA - J. Am. Med. Assoc., vol. 323, 2020, p. 1582–1589.
60.
ZHANG, B. et collab., « Treatment With Convalescent Plasma for Critically Ill Patients With Severe Acute Respiratory Syndrome Coronavirus 2 Infection », Chest, 2020, doi : 10.1016/j.chest.2020.03.039.
61.
LI, L. et collab., « Effect of Convalescent Plasma Therapy on Time to Clinical Improvement in Patients With Severe and Life-threatening COVID-19 » JAMA, 2020, doi : 10.1001/jama.2020.10044.
62.
MAROVICH, M., J. R. MASCOLA et M. S. COHEN, « Monoclonal Antibodies for Prevention and Treatment of COVID-19 », JAMA, 2020, doi :10.1001/jama.2020.10245.
63.
MARSTON, H. D., C.I. PAULES et A. S. FAUCI, « Monoclonal antibodies for emerging infectious diseases - Borrowing from history », New England Journal of Medicine, vol. 378, 2018, p. 1469–1472.
64.
MULANGU, S. et collab., « A randomized, controlled trial of Ebola virus disease therapeutics », N. Engl. J. Med., vol. 381, 2019, p. 2293–2303.
65.
CAO, Y. et collab., « Potent Neutralizing Antibodies against SARS-CoV-2 Identified by High-Throughput Single-Cell Sequencing of Convalescent Patients’ B Cells », Cell, vol. 182, 2020, p. 1–12.
66.
ROGERS, T. F. et collab., « Isolation of potent SARS-CoV-2 neutralizing antibodies and protection from disease in a small animal model », Science, 2020, doi : 10.1126/science.abc7520.
67.
PLOTKIN, S. A., « Correlates of protection induced by vaccination », Clinical and Vaccine Immunology, vol. 17, 2010, p. 1055–1065.
68.
GOWIN, E. et collab., « Does vaccination ensure protection? Assessing diphtheria and tetanus antibody levels in a population of healthy children:A cross-sectional study », Medicine, vol. 95, e5571, États-Unis, 2016.
69.
MOORE, S. M. et collab., « Rabies virus antibodies from oral vaccination as a correlate of protection against lethal infection in wildlife », Trop. Med. Infect. Dis., vol. 2, 2017.
70.
COUDEVILLE, L. et collab., « Relationship between haemagglutination-inhibiting antibody titres and clinical protection against influenza: Development and application of a bayesian random-effects model », BMC Med. Res. Methodol., vol. 10, 2010. 72. LAL, H. et collab., « Efficacy of an Adjuvanted Herpes Zoster Subunit Vaccine in Older Adults », N. Engl. J. Med., vol. 372, 2015, p. 2087–2096.
1.
73. FLYNN, J. A. L. et collab., « An essential role for interferon γ in resistance to mycobacterium tuberculosis infection », J. Exp. Med., vol. 178, 1993, p. 2249–2254.
2.
74. YU, J. et collab., « DNA vaccine protection against SARS-CoV-2 in rhesus macaques », Science, eabc6284, 2020, doi : 10.1126/science.abc6284.
3.
75. ADAMS, E. R. et collab., « Evaluation of antibody testing for SARS-Cov-2 using ELISA and lateral flow immunoassays », medRxiv, 2020, http://medrxiv.org/lookup/doi/10.1101/2020.04.15.20066407, doi : 10.1101/2020.04.15.20066407.
4.
76. NEWTON, A. H., A. CARDANI et T. J. BRACIALE, « The host immune response in respiratory virus infection: balancing virus clearance and immunopathology », Seminars in Immunopathology, vol. 38, 2016, p. 471–482.
5.
77. MEHTA, P. et collab., « COVID-19: consider cytokine storm syndromes and immunosuppression », The Lancet, vol. 395, 2020, p. 1033–1034.
6.
78. GUAN, W. et collab., « Clinical Characteristics of Coronavirus Disease 2019 in China », N. Engl. J. Med., vol. 382, 2020, p. 1708–1720.
7.
79. WANG, D. et collab., « Clinical Characteristics of 138 Hospitalized Patients with 2019 Novel CoronavirusInfected Pneumonia in Wuhan, China », JAMA - J. Am. Med. Assoc., vol. 323, 2020, p. 1061–1069.
8.
80. YANG, X. et collab., « Clinical course and outcomes of critically ill patients with SARS-CoV-2 pneumonia in Wuhan, China: a single-centered, retrospective, observational study », Lancet Respir. Med., vol. 8, 2020, p. 475– 481.
9.
81. MURTHY, S., C. D. Gomersall et R. A. Fowler, « Care for Critically Ill Patients with COVID-19 », JAMA - Journal of the American Medical Association, vol. 323, 2020, p. 1499–1500.
10.
82. CLARK, A. et collab., « Global, regional, and national estimates of the population at increased risk of severe COVID-19 due to underlying health conditions in 2020: a modelling study », Lancet Glob. Heal., vol. 0, 2020, p. 1– 15.
11.
83. TAY, M. Z., C. M. POH, L. RÉNIA, P. A. MACARY et L. F. P. NG, « The trinity of COVID-19: immunity, inflammation and intervention », Nature Reviews Immunology, vol. 20, 2020, p. 363–374.
12.
84. HUANG, Y. et collab., « Clinical characteristics of 36 non-survivors with COVID-19 in Wuhan, China », medRxiv, 2020, https://www.medrxiv.org/content/10.1101/2020.02.27.20029009v1, doi : 10.1101/2020.02.27.20029009.
13.
85. XIANG, J. et collab., « Potential biochemical markers to identify severe cases among COVID-19 patients », medRxiv, vol. 19, 2020, http://medrxiv.org/lookup/doi/10.1101/2020.03.19.20034447.
14.
86. FEI, J. et collab., « Reduction of lymphocyte at early stage elevates severity and death risk of COVID-19 patients: a hospital-based case-cohort study », medRxiv, 2020, doi : 10.1101/2020.04.02.20050955.
15.
87. YIP, M. S. et collab., « Antibody-dependent infection of human macrophages by severe acute respiratory syndrome coronavirus », Virol. J., vol. 11, 2014, 82 p.
16.
88. LIU, L. et collab., « Anti-spike IgG causes severe acute lung injury by skewing macrophage responses during acute SARS-CoV infection », JCI insight, vol. 4, 2019.
17.
89. TAYLOR, A. et collab., « Fc receptors in antibody-dependent enhancement of viral infections », Immunol. Rev., vol. 268, 2015, p. 340–364. ]
18.
90. HORBY, P. et collab., « Effect of Dexamethasone in Hospitalized Patients with COVID-19: Preliminary Report », medRxiv, 2020, doi : 10.1101/2020.06.22.20137273.
Cet article fut initialement publié dans le Globe and Mail le 6 juillet 2020.



